
Dans les chapitres 2 et 4, les formateurs vous présenteront les outils R et R Studio et vous montreront, via des vidéos pas à pas, la manière d'utiliser ces outils et de réaliser des analyses de données RNA-seq. Si vous souhaitez reproduire ces exercices chez vous, deux options sont possibles :
- Utiliser le compte qui vous a été créé sur les serveurs de l'Institut Français de Bio-informatique : l'IFB met en effet à disposition ses ressources pour toute la durée de la formation, notamment une instance R Studio. Utiliser le R Studio de l'IFB pour la formation vous assure d'avoir un environnement contrôlé et opérationnel, tous les package nécessaires au cours étant déjà installés. Veuillez noter que les comptes IFB qui ont été créés pour la formation sont temporaires. Ils seront désactivés fin 2024 et le projet associé avec cette formation sera supprimé. Vous pourrez continuer à travailler en local sur votre ordinateur après installation d'un R Studio localement, ou reprendre contact avec l'IFB à l'issue de la formation pour demander la création d'un "vrai" projet.
- Installer R et R Studio sur votre ordinateur : lors du chapitre 2 de cours, l'une des leçons sera dédiée à l'installation de R et R Studio. Vous pourrez ainsi réaliser les analyses proposées dans les chapitres 2 et 4 sur les données test fournies par les formateurs, ou bien encore sur vos propres données. Pour pouvoir reproduire les exercices proposés, votre ordinateur devra idéalement disposer d'un processeur i7 1.90 GHz, 8Go de RAM et 20Go d'espace disque. Il vous faudra installer, dans R Studio, les packages DESEq2, parathyroidSE, debrowser et ggplot2 ainsi que leurs dépendances.
Utilisation du R Studio de l'IFB
Se connecter à R Studio
Un mail de demande d'activation de votre compte vous a été envoyé début janvier, contenant un lien à cliquer pour activer votre compte. N'hésitez pas à regarder dans vos spams si vous n'avez rien reçu ! Une fois votre compte activé, vous recevrez un second mail automatique avec votre identifiant et mot de passe, qui vous permettront d'accéder au R Studio paramétré pour la formation.
Pour vous connecter à votre session RStudio, il faut passer via le lien suivant : https://ondemand.cluster.france-bioinformatique.fr. Lors de votre connexion, vous devrez "autoriser l'accès" de l'application OnDemand de l'IFB aux données de votre compte IFB ("Grant Access"). Vous pourrez ensuite sélectionner l'option "RStudio Server : core".

Dans le formulaire ensuite proposé, vous pourrez renseigner les paramètres suivants puis cliquer sur "launch" :
- R version : 4.1.1
- Account : 24_singlecell_canceropole
- Partition : fast
- Number of CPUs : 2
- Amount of memory : 8Go
- Number of GPUs : 0
- GPUs : none
- Number of hours : le nombre d'heure pendant lesquelles vous pensez utiliser R Studio aujourd'hui
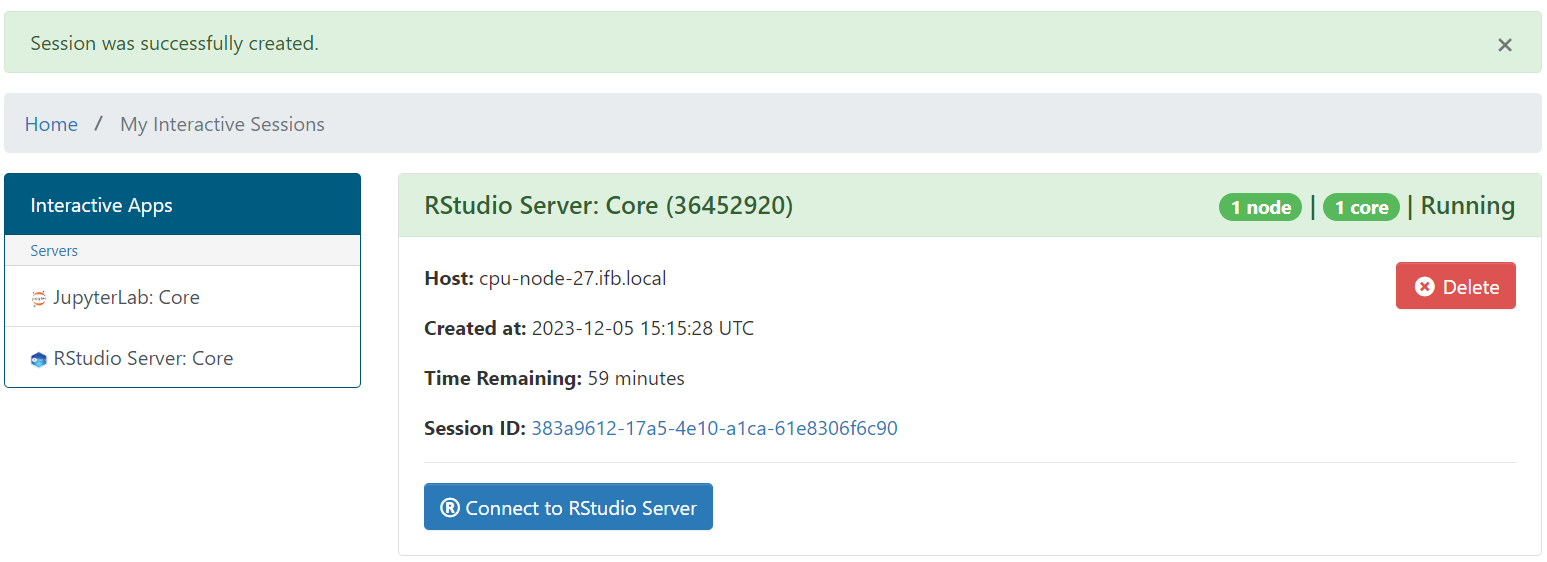
Une fois votre session créée (cela peut prendre quelques minutes), vous pourrez la lancer dans une nouvelle fenêtre / onglet en cliquant sur le bouton bleu "connect to RStudio Server". (si rien ne s'affiche après quelques minutes, rafraîchissez la page).
IMPORTANT : dans votre home se trouvent deux dossiers : "R" et "ondemand", ne les supprimez pas, ne les modifiez pas, c'est le contenu de ces dossiers qui permet de faire fonctionner R Studio.
Les fichiers .r et les données tests seront téléchargeables et fournis avec chaque cours, et vous pourrez les uploader sur R Studio dans votre dossier ~Home (qui sera votre espace de travail). Vous pourrez les uploader un par un au fur de votre apprentissage.
Enregistrer l'espace de travail
Vous l'avez compris, vous allez devoir recréer une session à chaque fois que vous recommencerez à travailler dans le R Studio de l'IFB. Pour retrouver votre espace de travail et reprendre là où vous vous en étiez arrêté lors de la session précédente, il est impératif de bien penser à sauvegarder son espace de travail à la fin de chaque session.
Pour cela, cliquez sur "save workspace as" dans la fenêtre "environnement" et enregistrez un fichier .Rdm dans votre home.
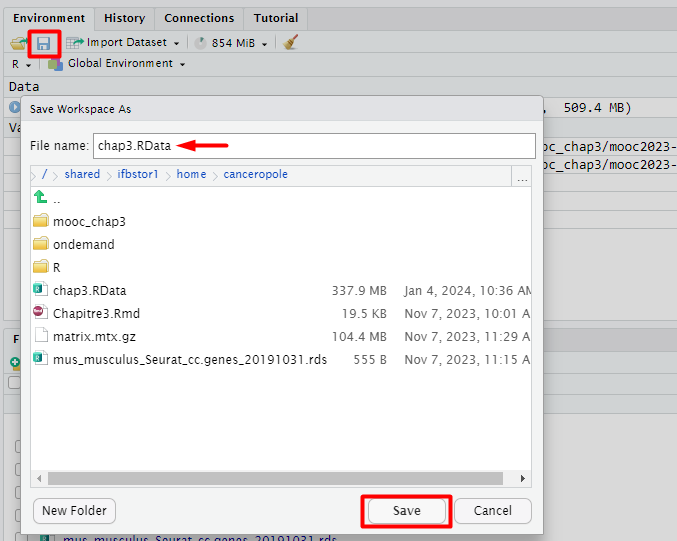
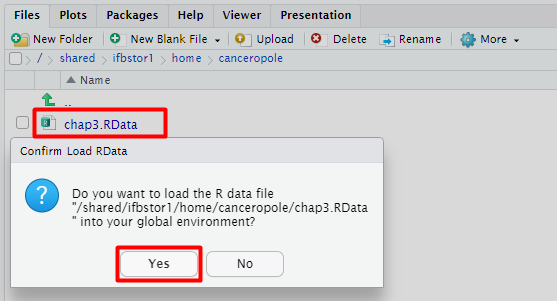
Il vous suffira, lors du lancement de la nouvelle session, de cliquer sur le fichier .rdm pour charger de nouveau toutes vos variables dans R Studio.
Travailler sur vos données
Si c'est la première fois que vous vous essayez à l'analyse de données RNAseq sur R Studio, nous vous conseillons de tester d'abord avec les données proposées par les formateurs avant de vous lancer dans l'analyse de vos données.
Si vous souhaitez essayer de mettre en pratique les exercices proposés d'analyse de l'expression différentielle sur vos données propres, il vous sera possible de les uploader sur votre compte R Studio. Veuillez noter que l'IFB ne dispose pas d'autorisation pour héberger des données de santé. Si vous souhaitez travailler sur des données de patients, merci de le faire uniquement à partir des tables de comptage, qui contiennent des données anonymisées.